导师:长健同学,欢迎你加入生殖大家庭。听你说对生信分析感兴趣,2017年同济大学高绍荣老师对不同阶段的小鼠胚胎打了[1] 质谱,蛋白组学分析中用到了R语言,你先学习下。
长健同学:老师,R语言去哪儿学习啊?
导师:B站上很多教学视频,简书、知乎上也有很多教程,还可以学习下之前的课件。加油!
一周后
长健同学:老师,我装了Rstudio,但还是不太懂文章里的各种分析的意义。
导师:没关系的,我带着你一步一步做,也稍微给你扩展一下。高老师这份蛋白组学数据已经进行了数据库搜索、质控,并且对生物学重复取了均值。基本的质控你需要掌握一些。拿到搜库结果首先要比较下各个样本组学中蛋白的数目,许多文章将蛋白过少的样本视为异常样本,结合具体情况进行剔除。之后要补充缺失值,方法挺多。这里主要给你介绍一下如何利用PCA进行数据降维和质控。为了给你举例,我根据高老师的数据编写了一份数据,供你行PCA分析,R包发给你了。
长健同学PCA供图
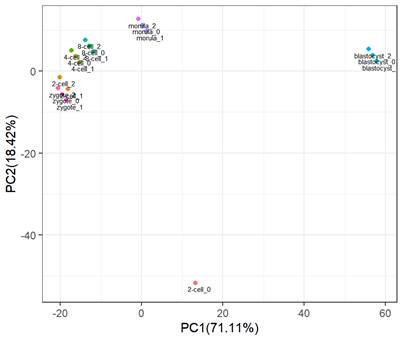
长健同学:老师,2-cell组的0号样本似乎离群了。
导师:是的,有什么疑问吗?
长健同学:老师,我虽然跑出来了,但不知道每个“包”是什么意思,更不知道要怎么用。
导师:“?”感兴趣的“包”就可以啦,比如“?prcomp()”,图像可以进一步优化。剔除异常样本后,再跑下PCA,看看生物学重复是否可靠,记得加置信椭圆。当分组样本较少时,R包无法给出PCA降维后的置信椭圆,一些文章会结合具体情况添加,这里为你介绍另外一种展示生物学重复和组间差异的方法——无监督聚类,Pheatmap包内置聚类,你先跑跑R包。
长健同学Pheatmap供图
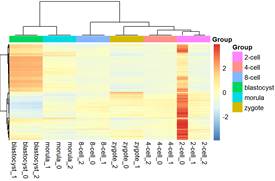
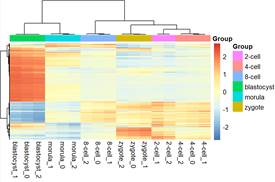
长健同学:老师,2-cell的0号样本确实离群了,其他样本的聚类挺符合预期的。
导师:是的,但数据的处理和热图的绘制需要你自己去研究和学习,比如数据中极值的处理、标准化和归一化,无监督聚类方法和热图色系的选择、其他标识的添加。多探索,多实践,很快你就能画出比较优秀的热图了。接下来学习下用Mfuzz包进行时间序列分析。
长健同学:好的老师。
导师:文章涉及了6个有序的胚胎发育节点的蛋白组数据,而Mfuzz包能够识别表达谱的潜在时间序列模式并进行蛋白聚类,结合富集结果,能发现一些生物学过程的变化情况。需要用某一蛋白的均值来代表某节点的该蛋白表达,取均值应考虑其代表性,特别是在样本聚类不是特别符合预期时。
长健同学:老师,我先跑跑R包。
长健同学Mfuzz供图
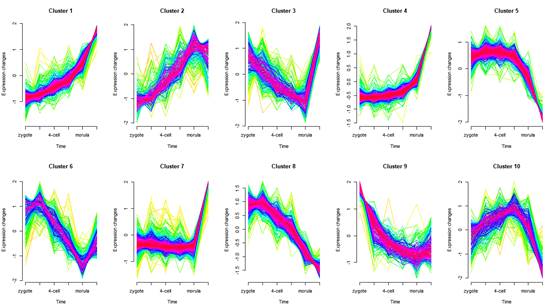
导师:建议你“?Mfuzz()”进一步优化结果图,而且每组可以挑选和标识出代表基因。接下来可以分别对每群的蛋白进行富集分析,结合具体情况,利用Pheatmap包、ggplot2包和AI等组合为大图展示。富集分析可以用R包进行,也可以使用Metascape等网站进行多数据库的富集分析。还有什么问题吗?
长健同学:老师,如果均值代表性不强,该怎样处理呀?
导师:此时Mfuzz包可能就不适用了。2022年沙倩倩老师衰老卵母细胞转录组数据的分析采用了另一种策略,即多个时间结点均与<30岁患者来源的年轻卵做比较,获得差异表达基因、分群并行富集分析,你可以去学习下。
长健同学:老师,我之前做年龄对妊娠结局影响的回顾性分析时,BMI这些指标往往在组间不齐,需要采用Logistic回归或PSM进行校正或匹配,如果组学分析时发现这些问题怎么处理啊?
导师:可是试试WGCNA包,多时间点分析时也可以用这个方法校正混杂因素、获得差异表达基因、分群并行富集分析。
长健同学WGCNA绘图

导师:很棒,快从输出结果里找出与年龄相关的模块和基因做富集分析吧。
长健同学:收到,谢谢老师!
彩蛋:感兴趣的同学可以联系赵涵老师研究生殷长健同学,索要文中示例数据及对应R包!(邮箱:17865195362@163.com)
参考文献
1.Gao Y, Liu X, Tang B, Li C, Kou Z, Li L, Liu W, Wu Y, Kou X, Li J, Zhao Y, Yin J, Wang H, Chen S, Liao L, Gao S. Protein Expression Landscape of Mouse Embryos during Pre-implantation Development. Cell Rep. 2017 Dec 26;21(13):3957-3969.
2.Wu YW, Li S, Zheng W, Li YC, Chen L, Zhou Y, Deng ZQ, Lin G, Fan HY, Sha QQ. Publisher Correction: Dynamic mRNA degradome analyses indicate a role of histone H3K4 trimethylation in association with meiosis-coupled mRNA decay in oocyte aging. Nat Commun. 2022 Jun 15;13(1):3441.